著者 Yo-Han KIM, Noriyuki TOJI, Keiichiro KIZAKI, Kei TAKEMURA, Shiro KUSHIBIKI, Shigeru SATO
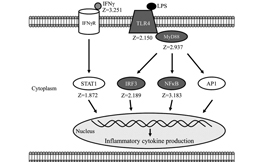
2019年に報告されたこの論文の著者らは、スターターのみを与えた子牛では、離乳後3週間時点のToll様受容体4によるシグナル伝達経路が活性化された一方、スターターと粗飼料を併給した場合には24時間平均ルーメンpHが高くなることを発見しました。スターターのみを与えた子牛ではルーメン上皮でのコレステロールの合成が低下した一方で、スターターと粗飼料を併給した場合にはエネルギー状態が高くなりました。
読む
著者 X. Song, P. P. J. van der Tol, P. W. G. Groot Koerkamp, and E. A. M. Bokkers
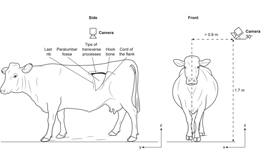
この研究では、乳牛の反芻胃(第一胃と第二胃)の運動性を、自動で遠隔解析するための、低コストの3D視覚システムを実演しました。この自動測定は、手動解析と同等の結果を示しました。従来の手法と異なり、この自動測定は非侵襲的で牛にストレスを与えません。
読む
著者 Robert D. Stewart, Marc D. Auffret, Amanda Warr, Alan W. Walker, Rainer Roehe, Mick Watson
本研究では、イルミナシーケンスやナノポアシーケンスによって得られた6.5テラバイトより多くのデータを用いて、包括的な解析を行いました。
読む
著者 H. M. Hammon, W. Liermann, D. Frieten, and C. Koch
初乳の給与時期が過ぎた後、子牛の体の成長と臓器の発達の可能性を活かすには、離乳前に少なくとも体重の20%のミルクを給与する強化哺乳が求められます。
読む
著者 D.C. Sockett
深部鼻咽頭ぬぐい液を用いた牛呼吸器病症候群(bovine respiratory disease complex :BRDC)の診断は、簡単で安全な技術として認められています。
読む
著者 Yifan Zhong, Mingyuan Xue, Jianxin Liu
この研究では、乳中体細胞数が高い乳牛では、特定のルーメン微生物の構成が変化している可能性が示唆されました。体細胞数 が多い乳牛は、乳量とルーメン内の揮発性脂肪酸濃度が低いことが示されました。しかし体細胞数が少ない乳牛と比べると、ルーメン細菌の多様性が高いことが分かりました。
読む
著者 Elie Jami, Adi Israel, Assaf Kotser & Itzhak Mizrahi
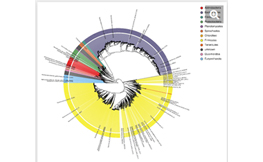
The ISME Journal誌に2013年に掲載されたこの論文では、生後からのルーメン生態系の変化について述べられています。
読む
著者 S. L. Gelsinger, W. K. Coblentz, G. I. Zanton, R. K. Ogden, and M. S. Akins
この研究では、スターター飼料の形状(ペレットまたはテクスチャライズ)が、全消化管またはルーメン内での各消化率(乾物、中性デタージェント繊維、でんぷん、窒素)に及ぼす影響について調査しています。得られた重要な知見の一つは、全消化管での見かけの消化率(特に乾物)は、週齢が大きくなるにつれて減少したことです。
読む
著者 Robert D. Stewart, Marc D. Auffret, Amanda Warr, Andrew H. Wiser, Maximilian O. Press, Kyle W. Langford, Ivan Liachko, Timothy J. Snelling, Richard J. Dewhurst, Alan W. Walker, Rainer Roehe & Mick Watson
この文献では43頭のハイランド牛から、913の細菌と古細菌のドラフトゲノムを示しました。 それらのゲノムの大多数は、今までに配列を解析されていない菌株と菌種の物でした。
読む
著者 M.R. Carvalho, F. Peñagaricano, J.E.P. Santos, T.J. DeVries, B.W. McBride, E.S. Ribeiro
本研究において研究者らは、7,500頭の泌乳牛を用いて分娩後早期の疾病が与える長期間の影響を評価しました。
読む